Einleitung
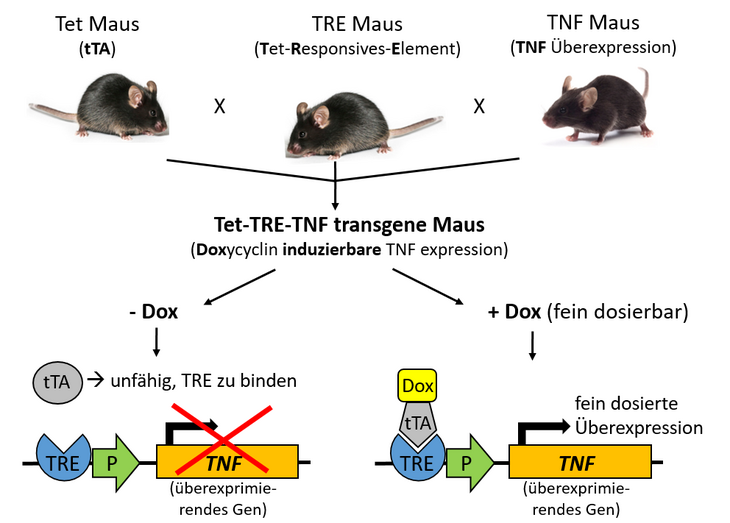
Im Rahmen psychischer Erkrankungen spielen inflammatorische Prozesse eine wichtige Rolle, die umweltbedingt aber auch genetisch prädisponiert aktiviert werden können. Neben experimentellen Daten aus Humanstudien spiegeln dies zunehmend Daten aus Tiermodellen zu depressionsähnlichem Verhalten wieder. Insbesondere ein Ungleichgewicht bestimmter inflammatorischer Marker z.B. aus dem Tumornekrosefaktor (TNF) Signalweg wird im Zusammenhang mit Depressionen und Angst bei Erkrankten beobachtet, genauso auch mit depressions- und angstähnlichem Verhalten bei Mäusen. Im Unterschied zum Mensch mit einer üblicherweise nur leichten TNF Level Erhöhung im Erwachsenenalter bei einer Gruppe depressiv Erkrankter weisen die bisher generierten transgenen Mausmodelle aber eine sehr starke TNF Überexpression auf und dies, anders als beim Mensch, vom Entwicklungsbeginn an. Solche artifiziellen Modelle erlauben keine gute Vergleichbarkeit zum Mensch, daher haben wir inzwischen ein transgenes Mausmodell mit Doxycyclin-induzierbarer fein-dosierbarer milder TNF-Überexpression entwickelt (s. Abb. oben). In diesem Modell fehlen bislang grundlegende vergleichende Untersuchungen zu individuellen alters- und geschlechtsbedingten physiologischen Änderungen im TNF- und weiteren (assoziierten) inflammatorischen Signalwegen. Ebenso fehlt der detaillierte Vergleich zwischen peripherem System, z.B. im Blut-Serum und zentralem System, z.B. in Hippocampus und Amygdala, die in engem Zusammenhang mit Angst und Depressionen stehen.
Ziel dieser experimentellen Doktorarbeit ist es daher, vergleichende Untersuchungen inflammatorischer Proteine im eigenen Tet-TRE-TNF Mausmodell durchzuführen.
Fragestellung
Dabei soll folgenden Fragen nachgegangen werden: Gibt es alter- und geschlechtsabhängige differenzielle Profile inflammatorischer Protein-Kandidaten? Spiegeln periphere Level inflammatorischer Protein-Kandidaten die Expressionen derselben Kandidaten in zentralen emotions-assoziierten Hirnregionen wieder?
Bearbeitung
Die Untersuchungen sollen an teils bereits vorhandenen Biomaterial-Proben von Mäusen unseres Tet-TRE-TNF Modells durchgeführt werden, teils an noch zu generierenden Proben. Je nach Material werden damit im Labor etablierte Assays nach gängigen Verfahren durch Quantifizierung von z.B. Proteinen und RNAs durchgeführt und die Daten anschließend statistisch analysiert.
Methoden
- Biomaterial-spezifisch, z.B. Fluorescence-encoded bead-based immunoassay (für Serum-basierte Proteine), RNA-isolation mit cDNA Synthese und qRT PCR (aus Blut und Hirn)
- Biomaterial-spezifische Analysen mit speziellen Softwares für z.B. serum-basierte Protein-Level Quantifizierung und differentiellen RNA-Expressionsbestimmungen
- R- und SPSS-basierte statistische Auswertung
Arbeitsprogramm
- Theoretische und praktische Einarbeitung in Hintergrund und Labormethoden
- Zusammenstellung und Analysen der Biomaterialien, Auswertung der Daten
- Anfertigung einer Dissertationsschrift