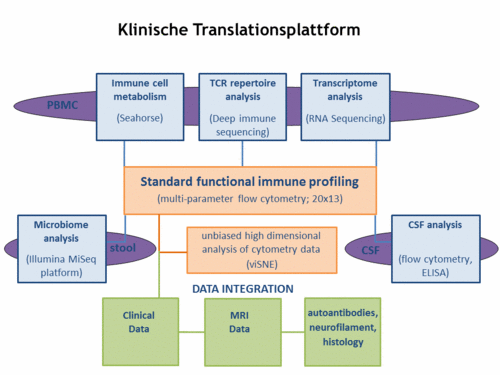
- Standardisierte funktionelle 13-Farben-Durchflusszytometrie mit multidimensionaler Analyse mittels verschiedener Algorithmen (verantwortlich: C. Groß und L. Klotz)
- Funktionelle Analyse des Immunzellmetabolismus, also mitochondrialer Atmungsaktivität, Glykolyse und Fettsäureoxidation (verantwortlich: L. Klotz)
- Analyse des T-Zell-Rezeptor-Repertoires mittels Sequenzierung (verantwortlich: L. Klotz, C. Groß und N. Schwab) RNA-Sequenzierungs-Plattform einschließlich single cell RNAseq (verantwortlich: Gerd Meyer zu Hörste)
- Analyse des Darm-Mikrobioms (verantwortlich: L. Klotz in Kollaboration mit dem Institut für Hygiene, UKM)
Ein besonderes Anliegen der Klinik für Neurologie ist eine enge Verzahnung von Klinik und Forschung. Um patientennahe Forschung auf hohem Niveau zu ermöglichen, wurde eine klinische Translationsplattform etabliert. Mit ihrer Hilfe können translationale Forschungsfragen anhand einer Vielzahl etablierter und standardisierter Methoden bearbeitet werden. Die Translationsplattform unterstützt die Konzeption und Durchführung wissenschafts-initiierter Forschungsprojekte an unterschiedlichen Patientenkohorten, die standardisierte Analyse von Patientenmaterial bei multizentrischen klinischen Studien sowie die Etablierung wissenschaftlicher Analysemethoden zur gezielten Beantwortung translationaler Fragen. Aktuell bearbeiten wir Forschungsprojekte zu unterschiedlichen neurologischen Krankheitsbildern wie der Multiplen Sklerose (MS), dem Schlaganfall, demenziellen Erkrankungen sowie Autoimmunenzephalitiden, einige davon im von der DFG geförderten Sonderforschungsbereich TR128 "Multiple Sklerose" und dem Kompetenznetz MS.
In den vergangenen Jahren haben wir eine Reihe wissenschaftsinitiierter Studien zur MS erfolgreich durchgeführt (z. B. die Studien ToFingo Successor, DIMAT und ALAIN). Zudem dient die Translationsplattform als zentrale Analyseeinheit von Proben aus den multizentrischen klinischen Studien TERIDYNAMIC sowie ENSEMBLE.
Innerhalb der Klinik für Neurologie arbeiten wir eng mit der neurologischen Biobank, dem Studienzentrum Neurologie sowie dem Liquorlabor zusammen.